Variational Autoencoder の実装 – Pyro(高速版)
概要
前回の記事では Pyro での VAE の実装例を紹介させて頂きましたが、コードは基本的に公式にあるコードを沿ったものになっていました。しかし、NumPyro との実行速度を比較する上では、画像をファイルから直接ロードするような Pyro のコードは若干不利になっていました。
そこで、今回は Pyro 版のコードを少しだけ修正して、画像を一旦全てメモリ上にロードしてから、処理を行うようなコードへ書き換えてみました。ちなみに、本家サイトのコードはこちらから見て頂くことができます。
Install Packages
!pip install pyro-pplImport Packages
import os
import gzip
import struct
import time
import numpy as np
import matplotlib.pyplot as plt
import torch
import torch.nn as nn
import torch.nn.functional as F
from torch.utils.data import Dataset, DataLoader
import pyro
import pyro.distributions as dist
device = 'cpu' # define 'cuda' for GPU
torch.device(device)plt.rcParams['font.size'] = 12
plt.rcParams['figure.figsize'] = [8, 6]Define Parameters
DATA_DIR = './'
Z_DIM = 50
HIDDEN_DIM = 400
OUT_DIM = 28 * 28
NUM_EPOCHS = 100
BATCH_SIZE = 256
LEARNING_RATE = 1.0e-3Define Dataset
前回からの変更点はこちらのみです。一旦、画像データを全てメモリ上にロードしてから、メモリ上のデータをカスタムのデータローダーで読み込ませるようにしています。
fname_train_images = os.path.join(DATA_DIR, 'train-images-idx3-ubyte.gz')
fname_train_labels = os.path.join(DATA_DIR, 'train-labels-idx1-ubyte.gz')
fname_valid_images = os.path.join(DATA_DIR, 't10k-images-idx3-ubyte.gz')
fname_valid_labels = os.path.join(DATA_DIR, 't10k-labels-idx1-ubyte.gz')def read_image(file):
with gzip.open(file, "rb") as f:
_, _, nrows, ncols = struct.unpack(">IIII", f.read(16))
data = np.frombuffer(f.read(), dtype=np.uint8) / np.float32(255.0)
return data.reshape(-1, nrows, ncols)def read_label(file):
with gzip.open(file, "rb") as f:
f.read(8)
data = np.frombuffer(f.read(), dtype=np.int8)
return datatrain_images = torch.from_numpy(read_image(fname_train_images)).clone().to(device)
train_labels = torch.from_numpy(read_label(fname_train_labels)).clone().to(device)
valid_images = torch.from_numpy(read_image(fname_valid_images)).clone().to(device)
valid_labels = torch.from_numpy(read_label(fname_valid_labels)).clone().to(device)def in_memory_data_loader(images, labels, batch_size=1, shuffle=False):
num_records = len(labels)
idxs = np.arange(num_records)
idxs = np.random.permutation(idxs) if shuffle else idxs
num_batch = num_records // batch_size
images = images.reshape(images.shape[0], -1)
images = (images > 0.5).to(torch.float32)
def fetch(k):
ret_idx = idxs[(k * batch_size):(k * batch_size + batch_size + 1)]
batch_image = images[ret_idx, ...]
batch_label = labels[ret_idx, ...]
return batch_image, batch_label
return num_batch, fetchnum_batch_train, train_fetch = in_memory_data_loader(train_images, train_labels, batch_size=BATCH_SIZE)
num_batch_valid, valid_fetch = in_memory_data_loader(valid_images, valid_labels, batch_size=BATCH_SIZE)Define VAE
class Decoder(nn.Module):
def __init__(self, z_dim, hidden_dim, out_dim):
super().__init__()
self.fc_dec1 = nn.Linear(z_dim, hidden_dim)
self.fc_dec2 = nn.Linear(hidden_dim, out_dim)
def forward(self, z):
x = self.fc_dec1(z)
x = F.softplus(x)
x = self.fc_dec2(x)
x = torch.sigmoid(x)
return xclass Encoder(nn.Module):
def __init__(self, z_dim, hidden_dim, out_dim):
super().__init__()
self.fc_enc = nn.Linear(out_dim, hidden_dim)
self.fc_loc = nn.Linear(hidden_dim, z_dim)
self.fc_log_std = nn.Linear(hidden_dim, z_dim)
def forward(self, x):
x = self.fc_enc(x)
x = F.softplus(x)
z_loc = self.fc_loc(x)
z_std = torch.exp(self.fc_log_std(x))
return z_loc, z_stdclass VAE(nn.Module):
def __init__(self, z_dim, hidden_dim, out_dim):
super().__init__()
self.encoder = Encoder(z_dim, hidden_dim, out_dim)
self.decoder = Decoder(z_dim, hidden_dim, out_dim)
self.z_dim = z_dim
def model(self, batch):
batch_dim = batch.shape[0]
pyro.module("decoder", self.decoder)
with pyro.plate("batch", batch_dim):
z_loc = batch.new_zeros((batch_dim, self.z_dim))
z_std = batch.new_ones((batch_dim, self.z_dim))
z = pyro.sample("z", dist.Normal(z_loc, z_std).to_event(1))
loc_img = self.decoder(z)
pyro.sample("obs", dist.Bernoulli(loc_img).to_event(1), obs=batch)
def guide(self, batch):
batch_dim = batch.shape[0]
pyro.module("encoder", self.encoder)
with pyro.plate("batch", batch_dim):
z_loc, z_std = self.encoder(batch)
pyro.sample("z", dist.Normal(z_loc, z_std).to_event(1))Inference
def train(svi):
loss_sum = 0.
for k in range(num_batch_train):
x, _ = train_fetch(k)
loss_sum += svi.step(x)
num_data = num_batch_train * BATCH_SIZE
loss_ave = loss_sum / num_data
return loss_avedef evaluate(svi):
loss_sum = 0.
for k in range(num_batch_valid):
x, _ = valid_fetch(k)
loss_sum += svi.evaluate_loss(x)
num_data = num_batch_valid * BATCH_SIZE
loss_ave = loss_sum / num_data
return loss_avepyro.clear_param_store()
vae = VAE(Z_DIM, HIDDEN_DIM, OUT_DIM).to(device)
optimizer = pyro.optim.Adam({"lr": LEARNING_RATE})
svi = pyro.infer.SVI(vae.model, vae.guide, optimizer, loss=pyro.infer.Trace_ELBO())%%time
losses = np.zeros(NUM_EPOCHS)
for k in range(NUM_EPOCHS):
t_start = time.time()
_ = train(svi)
losses[k] = evaluate(svi)
t_end = time.time()
print('Epoch {}: loss = {:.2f} ({:.3f} s)'.format(k, losses[k], t_end - t_start))Epoch 0: loss = 155.58 (2.507 s)
Epoch 1: loss = 131.54 (2.409 s)
Epoch 2: loss = 118.93 (2.395 s)
Epoch 3: loss = 109.64 (2.479 s)
## 中略
Epoch 97: loss = 81.31 (2.421 s)
Epoch 98: loss = 81.23 (2.408 s)
Epoch 99: loss = 81.18 (2.397 s)
CPU times: user 19min 48s, sys: 4min 11s, total: 24min
Wall time: 4min 1splt.plot(losses)
plt.xlabel('Epoch')
plt.ylabel('Loss')
plt.tight_layout()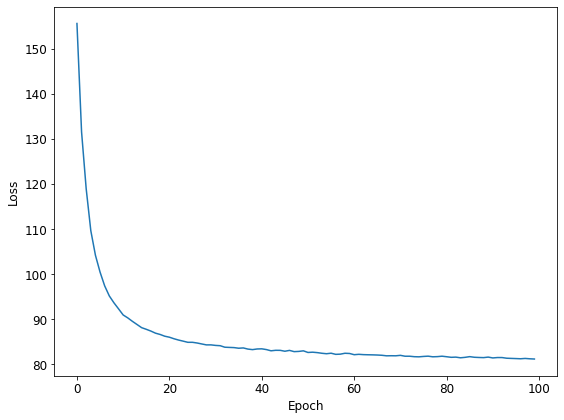
Summary
前回と同様に推論にかかった時間を計測しました(%%time で時間を測っているセルのところ)。ファイルから直接データをロードするよりは、かなり速くなりました。
GPUなし / Local Machine (Intel Core i5-8400) : 4min 1s
GPUあり / Google Colab (Tesla T4) : 2min 54s
GPUあり / Local Machine (Intel Core i5-8400 + Geforce GTX1070) : 1min 57s
前回は、データローダーにファイルから直接データをロードさせるような処理になっていましたが、それよりはかなり速くなりました。
ちなみに、前回の場合の実行時間はそれぞれ次の通りです。
GPUなし / Local Machine (Intel Core i5-8400) : 14min 4s
GPUあり / Google Colab (Tesla T4) : 13min 20s
GPUあり / Local Machine (Intel Core i5-8400 + Geforce GTX1070) : 5min 52s
ケースによりますが、2倍以上高速化していました。やはり、データローダーにファイルから直接データをロードさせていたのが、ボトルネックになっていたようです。比較的小さめなデータセットだったら、全てメモリ上に上げてしまった方が実行速度は速くできるのかもしれません(もしかしたら、データローダーの先読みの機構などのオプションをもっとうまく使えば、そんなことをしなくても高速化できるのかもしれませんが…)。
前々回紹介した NumPyro の場合は更に速いのですが、実はデータの読み込みの部分にも JAX による高速化が効くような細工がしてあり、かなり高速化の方へ振ったコードになっていました。よければ、そちらの記事もぜひご覧になって頂ければと思います。
※ なお、実行時間については、あまり正確には測っていませんので、あくまで参考程度に留めておいて頂けましたら幸いです。
お願い
記事につきましては、間違いないように十分に気をつけて書いたつもりなのですが、どこかで変なことを行っているかもしれません。お気付きの点がありましたら、ご面倒でもお問合せの方からご指摘頂けますと、ありがたいです。